平台包括了Heliscope单分子测序仪,世界上第一台DNA显微镜,Heliscope分析引擎,用于实时图像处理,专有的流式反应腔(flow cells),及单分子级试剂盒。
Helicos BioSciences公司以病毒M13为实验对象,首先将它的基因组截成小的片断,用一种酶将短小DNA标签附于每个片断的末端,在适当的位置锚定DNA片断。之后加入DNA复制酶和带有荧光标签的碱基或碱基对。当荧光DNA形成链时,就用相机拍下每个新加上的碱基对。
这一新方法称为“合成测序”,原则上与其它一些方法相同。不同之处在于,其它一些方法需要同时测序数千个相同的基因组片断以使信号足够“明亮”,新方法能够侦测到单个碱基的荧光。
HeliC0s生物科学公司——第三代基因测序技术开发者,已经利用HeliscoPe单分子基因测序仪对首例人类个体基因组进行单分子测序研究。
2009年8月10日发表在 ((Nature Biotechnology 上的一篇论文中对此方法进行描述,并且透露所使用的样本DNA来自于HelicOS公司 技术创始人Stephen Quake博士。
Quake博士是继James Watson先生和Craig Venter先生之后第三位DNA测序的非 匿名样本个体。Watson先生和Venter先生分别带领“人类基因组计划”和C elera公司两大团队对公布于2001年的第 一个人类基因组草图进行阐述。
如果目前基因组测序费用的最大目标是1000美元/人,那Helieos公司离这目标还很遥远,只是试剂就要花费约48000美元,这还不包括对测序仪本身的投资费用——接近 100万美元。
Helicos公司总裁SteDhen Lombardi先生告诉 Instrument News,Helicos公司测序技术发展的重要意义 在于此次基因测序工作是借助于斯坦福癌症研究中心独立 的基因组实验室,用Helic0s公司测序仪在2周内完成的, 而没有使用测序中心的一整套基础设施。
Helicos公司单分子测序仪的技术原理是利用合成测序 理论,将样本DNA数以百万的单链分子绑定在该仪器特有 的、没有背景荧光的玻璃表面,通过加入荧光标记的核昔 酸(一次加入4种核苷的1种)和聚合酶到单分子阵列中, 核苷酸会结合到DNA分子上特异性结合的位点上。
用激光 激发结合在D N A分子上的荧光标记的核苷酸,使标记物发 出荧光,相机以15ms速度快速扫描整个阵列,检测特异性 结合到D N A片断上的碱荧光基。在此之后,结合的核苷酸 对会被移动除去,然后,通过重复加入标记的核苷酸来重 复这~过程。 与Illumina/Solexa公司和Life Technologies/ABI公司 的第二代基因测序仪一样,Heliscope单分子基因测序仪对 很短的DNA片段也能读出来。与Illumina公司仪器一次读 100个碱基相比,Helicos公司限制可读的DNA片段平均为 32个碱基,因为D N A片段超过70个碱基就要考虑提高它的 技术工艺。 然而,Helicos公司生命科技的基因系统部门的科学运 营资深总监Kevin McKernan先生说明:利用Helicos公司 的测序技术可以达到96% 99%。这是由于收集大分子的基 因片段是非常困难的,测序时读取片段越短,这个工作的 困难越大
DNA测序技术已广泛应用于生物学研究的各个领域,很多生物学问题都可以借助高通量DNA测序技术予以解决。过去三年,大规模平行测序平台(massively parallel DNA sequencing platform)已经发展为主流的测序技术,这项测序技术的出现不仅令DNA测序费用降到了以前的百分之一,还让基因组测序这项以前专属于大型测序中心的“特权”能够被众多研究人员分享。目前,新的测序技术及手段还在不断涌现,比如最新的进展就包括建立序列数据库、建立序列数据分析新方法以及设计测序试验等等。新一代DNA测序技术有助于人们以更低廉的价格,更全面、更深入地分析基因组、转录组及蛋白质之间交互作用组的各项数据。今后,各种测序将成为一项广泛使用的常规实验手段,这有望给生物学和生物医学研究领域带来革命性的变革。
DNA测序技术经历了漫长而曲折的发展历程。迄今为止,我们获得的绝大部分DNA序列都是基于Sanger测序法获得的。在过去5年间,人们至少从以下四个方面刺激了DNA测序技术的发展(表2)。
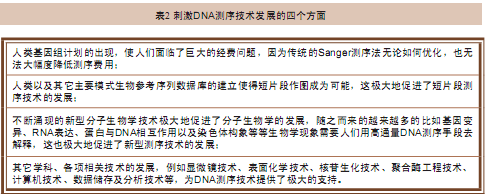
1. 具有代表性的新一代DNA测序仪
最近市面上出现了很多新一代测序仪产品,例如美国Roche Applied Science公司的454基因组测序仪、美国Illumina公司和英国Solexa technology公司合作开发的Illumina测序仪、美国Applied Biosystems公司的SOLiD测序仪、Dover/Harvard公司的Polonator测序仪以及美国Helicos公司的HeliScope单分子测序仪。所有这些新型测序仪都使用了一种新的测序策略——循环芯片测序法(cyclic-array sequencing),也可将其称为“新一代测序技术或者第二代测序技术”。
所谓循环芯片测序法,简言之就是对布满DNA样品的芯片重复进行基于DNA的聚合酶反应(模板变性、引物退火杂交及延伸)以及荧光序列读取反应。2005年,有两篇论文曾对这种方法做出过详细介绍。与传统测序法相比,循环芯片测序法具有操作更简易、费用更低廉的优势,于是很快就获得了广泛的应用。
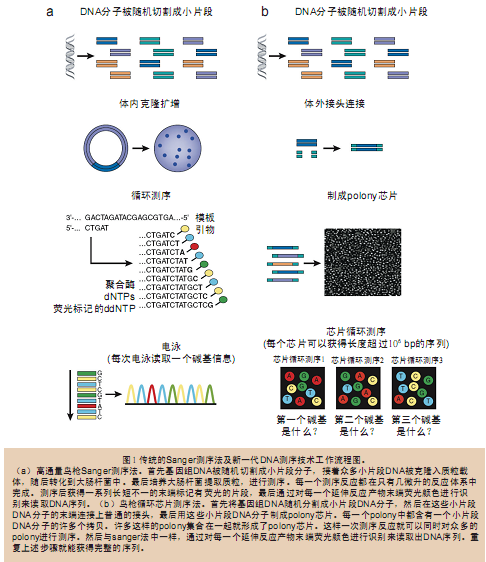
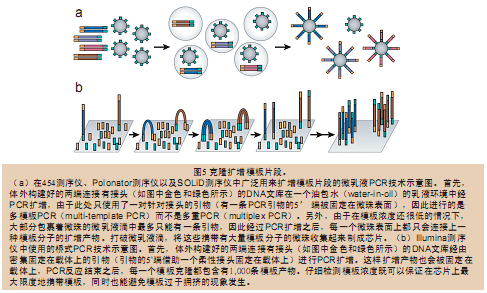
虽然这些新一代测序仪以及芯片的实际制作过程似乎都和传统的测序方法有很大的不同,而且各有特点(表3),但实际上它们背后的原理和技术都是非常相似甚至是相同的(图1b)。新一代测序法首先也是将基因组DNA随机切割成小片段DNA分子,然后在体外给这些小片段分子的末端连接上接头制成文库,也可以使用配对标签(mate-paired tag)制成跨步文库(jumping libraries)。随后可以通过原位polony(in situ polony,小词典1)、微乳液PCR(emulsion PCR)或桥式PCR(bridge PCR)(图5)等方法获得测序模板。
上述方法有一个共同点,那就是任何一个小片段DNA分子的PCR扩增产物都是在空间上聚集的:原位polony法和桥式PCR法中所有的产物都集中在平板的某处,在微乳液PCR法(emulsion PCR)中所有的产物都集中在微珠的表面。真正的测序反应本身和传统测序法一样,是由重复的聚合酶促反应和最后的荧光读取分析反应组成(图6)。本文讨论的所有测序仪都是使用合成测序法(sequencing by synthesis),即通过聚合酶或连接酶不断地延伸引物获得模板序列,最后对每一轮反应的结果进行荧光图像采集、分析,获得序列结果。

注:虽然目前测序片段长度短和准确率不高这两个缺点限制了新一代测序技术的应用,不过应该坚信,我们最终一定会克服这些问题。就好像经过了30年的努力,传统的测序技术也今非昔比,到达了今天的水平一样。
1.1 454测序仪
454测序仪的出现极大促进了测序业务的开展,科研人员已经将测序技术作为解决科研工作中许多常见问题的利器。这是因为454测序仪在以下几个方面取得了质的突破:首先是解决了高通量测序问题;其次它简化了样品准备步骤,将以往转化大肠杆菌扩增质粒的繁琐过程全部用简单的体外PCR扩增法替代了;最后,它缩小了测序反应体积,节省了试剂。这样,454测序仪做到了以极其低廉的价格进行大规模平行测序反应。它的测序规模之大、测序费用之低是以往的测序仪无法匹敌的。454测序仪与其它的新一代测序仪一起,降低了测序检测的费用,推动了测序技术平民化进程,使得小实验室也能开展测序检测项目,打破了以往只有少数几个大型测序中心才能进行测序研究的“垄断地位”。在过去的18个月里,由于有了454测序仪的帮助,人们对人类基因组的结构有了更深入的了解,同时第一次使用非Sanger测序法对个人进行了测序,还建立了一种发现小RNA的新方法。不过,要能让更多的人使用上新一代的测序产品,它们还需要变得更便宜,并且更加容易操作。在一段时间之内,454测序仪必定会进一步降低测序费用,帮助人们迎接个人基因组时代的到来。
自从诺贝尔奖得主Frederick Sanger和Walter Gilbert(图2)分别发明了链终止法DNA测序技术(sequencing by chain termination technique)和链断裂法DNA测序技术(sequencing by chain fragmentation technique)之后,人们就一直希望能够扩大DNA测序技术的处理规模。到了今天,我们对测序技术的需求和对计算机技术的需求一起出现了迅猛的增长,因为测序技术的发展速度已经远远跟不上实验要求的增长速度。于是出现了好几种替代Sanger测序法的新型测序方法,比如杂交测序法、借助原子力显微镜(atomic force microscopy)直接DNA成像测序法(direct imaging of DNA sequence)、质谱分析法、合成测序法以及微液流测序法等等。在我们进行人类基因组计划时还出现了三项技术改进方法,即使用荧光标记物取代了放射性标记物来标记终止碱基(双脱氧碱基);使用毛细管电泳(capillary electrophoresis)取代了传统的平板凝胶电泳;建立了末端配对测序法(paired-end sequencing)来对质粒、fosmid、人工细菌染色体(BAC)等短片段序列进行测序,解决了测序长度带来的限制问题。同时,开展研究的自动化液体分装技术(liquid-handling robotics)帮助我们摆脱了人工试管操作,可以用自动化的方式在微量滴定板(microtiter plate)上装载待测序样品(质粒等),极大地降低了测序的费用和劳动强度。

随着美国454 Life Sciences公司(该公司现已被美国罗氏公司收购)的第一台新一代测序仪——454测序仪的面世,我们获得了一种完全不同的测序方式。454测序仪引领的新一代测序技术在一直困扰传统测序技术的三个瓶颈问题上取得了突破。这三个问题分别是文库制备、模板制备和测序。而且,在随后出现的其它新一代测序仪产品身上,我们或多或少都会发现在454测序仪上使用到的技术,这也足以说明454测序仪的技术创新的确取得了巨大的成功。
454测序仪的先行者地位使它对整个测序业的影响远远超过了其它新一代测序仪竞争对手。这一点从Leamon、Rothberg等人撰写的一篇介绍2005年技术进展的论文被引用了570多次的事实,以及有100多篇经过同行审议的关于人类遗传学、代谢组学、生态学、进化学以及古生物学的论文(peer-reviewed publications)都是使用454测序仪开展的研究多个事实中都能够得到证明。454测序仪技术是继Sanger测序技术之后出现的第一个用于对细菌基因组进行从头测序的新技术,也是第一个被用来对人类基因组进行测序的非Sanger测序技术。其它使用454测序仪开展的重要研究项目包括探究蜜蜂消失原因的项目、研究人类基因组重排复杂性的项目、建立用于研究传染性疾病新方法的项目以及对尼安德特尔人(Neanderthal)基因组的测序项目等。
1.1.1 摩尔定律对454测序仪的影响
454测序仪的迅猛发展不是因为我们想要Sanger测序仪小型化,而是因为新型奔腾芯片的出现以及摩尔定律法则给我们带来的希望。很明显,常规的人类基因测序项目会对我们处理测序技术的能力提出更高要求,这与我们对计算机处理能力的要求是一样的。不过,只有将计算机的电子管换成晶体管,才为后来集成电路技术的发展提供了可能,这正是计算机产业发展的关键所在。而希望对传统的毛细管电泳技术进行改良,提高它的速度和处理规模,正如只用电子管直接制作集成电路一样不可能。因此,如果将各种测序技术比作一个个晶体管,将一系列测序步骤整合起来比作集成电路,那么也就可以用摩尔定律来预测DNA测序技术的发展速度了。
合成测序法概念虽然在提出的时候还不算成功,但它的出现为测序仪小型化奠定了基础。基于合成测序法出现了两种策略:一种是循环可切除终止测序法(cyclic reversible termination technology),即依次逐个添加荧光标记的碱基,继而检测荧光信号,切除荧光基团,如此往复;另一种策略是焦磷酸测序法(sequenced by detecting pyrophosphate release)。454测序仪采用的正是焦磷酸测序法,因为它似乎比第一种方法的效率更高。结果证明,454公司的选择是正确的。454测序仪采用的是小型化焦磷酸测序反应,测序模板准备和焦磷酸测序反应步骤都是在固态芯片上完成的。
实际上,早在上世纪90年代中期,焦磷酸测序技术就已经被科研界用来进行基因分型工作了,但那时的焦磷酸测序技术还不能够满足标准的测序实验要求,因为它的测序长度太短,因此只能用于旨在发现SNP的基因分型研究当中。当时进行基因分型操作时,是在微量滴定板(microtiter plate)上进行的,可以连续进行最多96次基因分型实验,平均每个样品花费20美分。那时焦磷酸测序还不能用于从头测序工作,因为从头测序需要对每一个尤其是第一个碱基都能准确地区分清楚,而焦磷酸测序只能简单地对已知位点的碱基进行检测,而且从头测序要求的测序长度也是焦磷酸测序法无法达到的。
不过,由于焦磷酸测序的原理是通过检测碱基掺入时发出的光来进行测序的(图3),所以它并不需要类似于电泳之类的物理分离过程来对碱基进行区分。这也就是说焦磷酸测序仪可以“缩小(减)”到只需要检测光线就够了,而不需要像传统的测序仪还需要电泳设备,而这正是限制传统电泳仪小型化的关键所在。发光检测方法还能够进行多路平行操作,但是直到454测序仪出现之前,还没有人这样做过,以前都是依次进行检测的。和晶体管早期的遭遇一样(当时人们也怀疑晶体管替代不了电子管),人们同时对高密度的,用于并行焦磷酸测序的反应也充满了疑问。不过,当我们不再在溶液中进行测序反应,而是将测序模板、所有的试剂(酶)都固定在平板上制成芯片之后,就获得了小型化的,能进行多路并行处理的测序仪,这就与晶体管被小型化并整合成
集成电路的过程一样。此外,借助微量滴定板上一个个的小孔所达到的将不同测序反应进行分隔这一目的,也能通过在单个固相支持物上进行严密包裹(隔离)的反应来实现。在这些各自隔绝的反应体系中,链聚合反应速度和发光速度都能通过对反应试剂和产物弥散状况进行严密的控制来进行精密的调整。
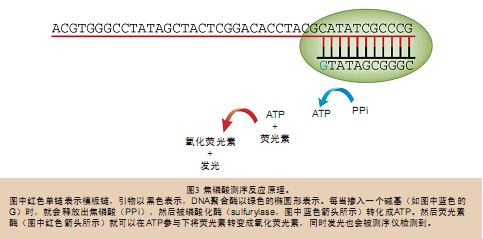
1.1.2 新的并行试验方法
在开发新型高通量、高并行运行方法时碰到的一个关键问题是,如何将反应试剂同时加入数量如此之多的各个反应体系中?在焦磷酸测序的过程当中需要反复加入不同的碱基以供测序反应使用,而当时的自动化加样设备无法有效地做到对这么多的反应体系同时循环加样。于是,开发一种全新的高密度并行处理方法这一重要课题又再一次摆在了科研人员的面前。这一次,我们找到了一个非常简单但是又很巧妙地方法。在高密度的反应芯片表面使用层流(laminar flow)加样方式,反应试剂会通过扩散作用很好地进入每一个反应体系,而且也可以用层流的方式洗去多余的反应试剂。现在,所有的新一代测序仪都采用了这种层流加样方法。
为了将每个单独的测序反应都分隔开来,我们一开始使用平板(芯片),不过在平板上平均每一平方厘米的面积上最多只能同时进行数百至数千个反应。但我们希望达到的是在每平方厘米的面积上同时进行100万个测序反应,这样才能令测序仪小型化,同时节省试剂并进行快速成像和测序。为了实现更高密度的测序反应,我们在平板上制作了很多小孔,将每个反应体系都安置在这些小孔中,这些小孔都足够深,足以分隔每个反应体系。虽然这种方法极大提高了测序反应的密度,缩小了平板的面积,但是要达到我们的要求还是需要60mm×60mm大小的芯片才行。
针对图像采集问题使用了商业化的天文学照相(astrological grade camera)器材,在电荷偶合装置(CCD)的表面连接上光纤束(fiber-optic bundle)。这些光纤是锥形排列的,这样可以将大范围的光信号都传输到CCD表面上很小的一个范围。采取下面两个步骤,我们就可以制成含有高密度小孔的芯片:先将光纤束连接到类似于载玻片一样的一次性芯片上,然后用酸蚀刻(acid etching procedure)技术在玻片的另一面打上小孔。这种酸蚀刻技术是根据制作生物传感器的技术改进而来的。
454公司制作的每张芯片上可以达到数百万个小孔,每一个小孔都是一个独立的“反应站”,互不干扰,测序反应发出的光被连接在芯片上的光纤传送到CCD记录下来(图4)。这种芯片就好像集成电路一样一次可以同时处理数百万个测序反应。这种芯片同样也能被其它通过发光检测技术的产品所使用。454测序仪也没有像以前的96孔板焦磷酸测序仪那样使用液态的试剂,而是将试剂和模板统统都吸附在一个个微珠上,然后把这些微珠一个个地放到芯片上的小孔中,每孔一个微珠。这种固定步骤不仅保证了每孔测序反应的独立性,也极大地节省了试剂消耗费用。
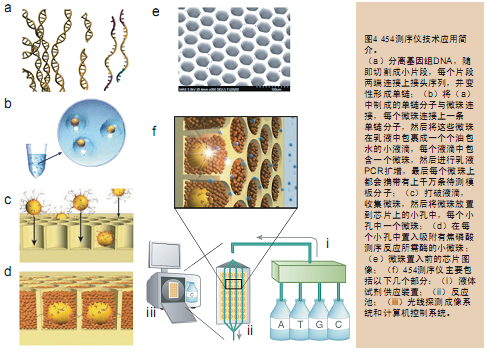
要想实现高通量基因组测序,只对测序步骤进行优化还是远远不够的。人类基因组计划花费的30亿美元经费中有很大一部分都用在了测序样品制备阶段。当时即使是采用最简单的制备样品方法也需要将目标片段克隆到细菌中,挑克隆,再转到96孔板,然后进行克隆扩增,提取质粒,制备测序模板。这种工作流程既耗时也耗钱。
如果采用新型的文库制备方法就可以极大地节省这部分开支,这种新型的方法是先分离基因组DNA,随机切割成小片段分子,然后通过有限稀释(limiting dilution)和聚合酶扩增反应,即体外克隆方式(clones without bacterial)制备模板片段。这样,从模板制备到最后的测序反应整个过程都能够在体外完成。
1.1.3 从发明到创新
从概念的提出到最后技术上的实现,454测序仪主要关注两个方面,首先是开发蚀刻光纤玻片;其次,改进焦磷酸测序方法使其能在固相支持物上进行,即将其改造成固态焦磷酸测序法,同时也对模板及文库构建方法进行了改进,让454测序仪能进行长片段测序工作和从头测序工作。
1.1.3.1 在蚀刻板上的小孔中进行固态、长片段焦磷酸测序反应
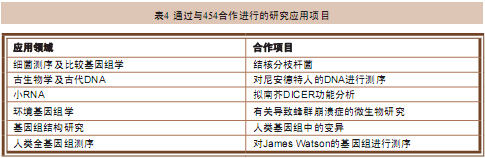
蚀刻技术经过改良之后能在75mm×75mm的玻片上刻出深55μm、宽44μm的小孔。而开发固态测序方法和改良测序长度则是两个紧密相关的问题,因为在固定的小孔中反应实际上就能改进测序质量和测序长度。由于反应试剂能迅速渗透到小孔中,因此反应速度也会加快。而且这里也没有使用三磷酸腺苷双磷酸酶(apyrase)提取未参与反应的碱基,而是将芯片置入反应池中通过层流液体的快速渗透作用将多余的未参与反应的碱基和反应副产品洗掉,由此得到100bp~500bp的测序长度。在能有效去除多余碱基的同时,每轮反应中聚合酶的效率也得到了极大提高。这样高效率的聚合反应使得454测序仪具有较长测序长度的同时也保证了高准确性,测序长度在200bp时的准确率高达99.5%。这是因为通过降低小孔中残存的未参与反应的碱基浓度,可以降低这些碱基对聚合酶活性的抑制作用,或者降低这些碱基导致的延后错误(carry-forward error,即由于未参与反应的碱基导致的测序反应不同步现象)的发生率。454测序仪在测序长度和准确率方面具有优势还因为其在应用流体学、表面化学和酶学(包括选择更好的聚合酶、在更高的温度进行测序反应以及更换及平衡各个酶组分)等方面都有创新(表4)。
还有一些能提高测序精度和测序长度的技术,不过暂时还没有商业化产品。这些技术包括使用可切除的终止碱基(reversible terminator)提高对同聚物(homopolymers)的检测精度;双末端测序法(double-ended sequencing),即同一模板的两条链均不测序;以及选择性酶固定法(alternative enzyme-immobilization method)等。这些技术改进还都没有用到测序仪产品中,有一部分原因是因为现在还没有必要使用。
注:蜜蜂群崩溃症(honeybee colony collapse),指的是来自养蜂业的蜂箱或自然界存在的欧洲蜜蜂群的工蜂突然消失的现象,又称作Colony collapse disorder(CCD)。
1.1.3.2 模板制备程序
完全的体外大规模模板制备工作是达成高通量、低价格测序技术的前提。已广泛使用的乳液PCR扩增技术就是一种很好的方法。不过,由于很难在热循环测序反应中保证乳液微滴的稳定性,因此最开始实验的模板扩增方法是恒温扩增法(isothermal)。
乳液PCR不需要借助细菌的帮助就能扩增模板,虽然这一点非常诱人,但最开始时并没有合适的表面活性剂能帮助乳液在热循环过程中保持稳定。于是出现了恒温扩增法,即滚环扩增反应(RCA)。虽然滚环扩增反应的产量非常高,但这些产物中大部分都不能用来作为测序模板。因此,还需要找到一种不需要细菌扩增,能用于有限稀释的模板扩增新方法。于是,人们又把目光转回了PCR法。在RCA法中,首先将模板克隆有限稀释之后置入光纤玻片上的小孔中,然后用橡胶衬垫把光纤玻片封闭起来,将玻片放入传统的平顶PCR仪进行扩增。这种方法取得了成功,但是效率不高,因为在玻片中的热质量(thermal mass)和它的钳效应(clamping mechanism)需要更长的PCR循环时间,而且模板的有限稀释度不能低于10%。孔与孔之间的相互污染现象也是一个不容忽视的问题。不过无论如何,该方法还是第一个首先从全基因组文库中扩增模板然后使用非Sanger、非Gilbert测序法对基因组进行从头测序的方法,也是第一个使用体外模板扩增技术进行全基因组(腺病毒基因组)测序的方法。
乳液滴的热稳定性问题最终通过加入用于制造炸药的表面活性剂得到了解决,于是乳液PCR技术马上在众多新一代测序仪中得到了广泛的应用。因为乳液PCR技术具有高效性、可扩展性,既能从30Kb的腺病毒基因组中扩增模板,也能从好几Mb的肺炎链球菌(Streptococcus pneumoniae)基因组中扩增模板。
随着测序精度、测序长度、乳液滴稳定性等各方面技术的不断发展,454测序仪已经不仅仅用于对细菌级别的基因组进行测序了,还能对更高级、更复杂的生物基因组进行测序,例如现代人类基因组、尼安德特人基因组以及环境基因组等。
1.1.3.3 文库制备
文库制备包括以下几个步骤,首先随机切割样品基因组,获得大量DNA片段,然后接上接头进行扩增反应。454测序仪的样品制备程序和Craig Venter等人的鸟枪法样品制备程序有着本质的差别。454公司采用的是如图4中所示的有限稀释、乳液PCR扩增法,而没有鸟枪法中的细菌克隆繁殖步骤。去掉了细菌繁殖步骤极大地提高了整个测序工作的速度和效率,同时避免了由于细菌繁殖导致的序列丢失的可能性。这种方法同样对古老DNA和代谢基因组学的研究也非常适用。末端配对文库制备方法的建立同样帮助454测序仪获得了对复杂基因组从头测序、对重复片段测序以及对基因组结构(复制、重排)展开系统研究三种能力。这种末端配对文库的制备方法是受到了Bender科研小组对果蝇(Drosophila)制备跨步文库(jumping library)方法的启发而发展得来的。
1.1.4 应用范围
随着越来越多重要的研究领域受到测序技术的影响,454公司开始和其它商业和学术机构开展合作,进行样品测序和分析工作。这些合作项目又进一步验证了454测序仪使用的技术能够在众多领域中发挥作用,例如末端配对文库技术对于研究基因组结构的作用和乳液PCR技术捕获目的DNA片段的作用等。
1.1.4.1 细菌基因组测序和比较基因组研究
为了测试454测序仪在全基因组测序方面的能力,454公司一开始就参与了一项合作项目,该研究项目会对4株结核分支杆菌基因组进行测序,这四株结核分支杆菌分别是一株对R207910具有耐药性的结核分枝杆菌(Mycobacterium tuberculosis)菌株,基因组大小约4Mb;两株对R207910具有耐药性的耻垢分支杆菌(Mycobacterium smegmatis),基因组大小约6Mb;以及一株正常的耻垢分支杆菌(Mycobacterium smegmatis),基因组大小约6Mb。他们希望能发现结核分枝杆菌(Mycobacterium tuberculosis)对R207910产生抗药性的机制。该项研究清晰的展现了454测序仪在测序速度和测序精度方面的优势。使用传统的Sanger测序法对一个4Mb的基因组和3个6Mb的基因组进行测序需要好几个月的时间,而用454测序仪,在只有一位实验人员参与实验的情况下,包括样品制备等步骤在内所用的时间仅需要一周。而且使用454测序仪还避免了传统测序方法中细菌克隆阶段可能出现的错误,获得了高质量的测序结果,发现了导致结核分枝杆菌对R207910产生抗药性的两个点突变位点。这项研究成果让我们在最近的40年内第一次找到了特异性治疗结核病的药物,同时也对454测序仪在细菌基因组测序方面的应用价值有了深刻的体会。随后,454测序仪又参与了比较基因组学研究项目、对高致病性细菌空肠弯曲菌(Campylobacter jejun)基因组的从头测序项目、对幽门螺杆菌(Helicobacter pylori)在慢性胃炎致病过程中的进化研究项目、从南极海冰细菌(Antarctic sea ice bacterium)中新发现冰结合蛋白(ice-binding protein)并对其测序的研究项目,以及在引起肺炎、脑膜炎和泌尿道感染的细菌中发现致病因素的研究项目等。
由于454测序仪不会因为细菌克隆产生测序误差,所以在对结核分枝杆菌抗药性的研究中表现出了非常强的发现突变位点的能力,这一点也被后来的其它研究项目所证实。此外,最近在用454测序仪进行的人类基因组测序项目中发现了长达29Mb的片段与人类基因组参考序列build-36不相符,这些片段被认为是参考序列中不存在的序列,属于基因组中的常染色质部分。不过,还需要注意的是,有些报道称由于重复片段的存在会出现序列组装错误,而且小模板片段雾化(nebulization)处理这种方式也会造成测序错误出现。
1.1.4.2 小RNA测序
对于包括miRNA在内的小RNA的研究兴趣从2005年开始就持续不断升温,而2005年恰好也是454测序仪上市的那一年。454测序仪以其不需要进行传统的细菌克隆步骤和足以覆盖只有21bp长的miRNA的测序长度等优势,很快就在miRNA的作用研究之中占据了一席之地。454测序仪最早参与进行的miRNA研究是对拟南芥(Arabidopsis thaliana)miRNA开展的研究。随后马上又参与了另一项研究项目,在这个项目中我们在小鼠体内发现了一种新型的小RNA——piRNA。这些研究项目为我们在人类、黑猩猩、斑马鱼和肿瘤细胞系中开展小RNA研究铺平了道路。454测序仪具有的这种对小RNA进行研究的能力使它在众多有关RNA的研究领域都能有所作为,例如转录体研究领域、EST研究领域、5’-RATE研究领域和基于转录体的SNP研究领域等。
1.1.4.3 在古生物学和古DNA研究领域的作用
要用传统的测序方法对尼安德特人的基因组进行测序研究非常困难,因为这些古老DNA量非常少,而且都早已裂解成了片段。一开始,454公司使用比较容易得到的不太重要的古代DNA样品检验了454测序仪对它们的测序能力,结果非常好,尽管当时454测序仪的测序长度只有100bp。不过,尼安德特人的基因组片段长度基本上都介于40bp~90bp之间,而且最近开发的乳液PCR方法也能够对微量(单分子)样本进行很好的扩增。于是,454测序仪参与了对38,000年前古老的尼安德特人的基因组进行测序的工作,研究结果分别发表在了好几篇论文当中,引起了广泛的关注,并促进了古生物学基因组的研究。随后有人对长毛象(woolly mammoth)和更新世狼(Pleistocene wolves)的基因组开展了测序研究。
1.1.4.4 环境基因组学和感染性疾病研究领域
美国在2001年爆发了炭疽恐怖袭击危机之后,454公司便对如何使用454测序仪对复杂的、未知的、未人工培养的环境微生物基因组进行测序展开了研究。前后两个合作研究项目均表明454测序仪能够用于从DNA混合样品中发现未知微生物并对其进行分类。在第一个研究项目中,有三名患者都接受了同一名澳大利亚器官捐赠者的器官,之后均因不明原因而死亡。从这三名死者身上提取了非人类DNA样品进行测序,结果获得了144,000条序列。分析后发现,这些序列分别属于一种沙粒病毒科(Arenaviridae)家族病毒的14个不同基因。随后进行的第二项研究在对健康蜂群和患病蜂群进行环境基因组学比较研究之后发现,以色列急性麻痹病毒(Israeli acute paralysis virus)是导致蜜蜂蜂群崩溃症的元凶。上述这些研究都突出了454测序仪的一个特点,即在样品准备前不需要进行克隆或预扩增步骤,因此非常适用于对未知的未能人工培养的物种进行测序。这些特点也在其它对地下矿藏、深海、土壤和高盐等环境下进行的环境微生物构成方面的研究所证实。
1.1.4.5 基因组结构研究领域
454测序仪技术的进步使它能够适用于更多的科研领域。最新开发的末端配对测序法(paired-end sequencing)就非常适合用于发现人类基因组当中的结构变异。末端配对作图过程(paired-end mapping),简单来说就是对一个非洲人和一个欧洲人的基因组进行测序后发现结构变异并对其作图,最终将1,000多个3Kb或更长的结构变异片段定位到人类基因组参考序列中。研究发现,在人类基因组当中存在的结构变异远远超过了人们的预计,其中有很多变异都会造成非常重要的表型改变。这项对诺贝尔奖得主James Watson基因组进行测序的项目和其它相关研究,一起使得“人类基因多样性(human genetic variation)”这一科学命题成为了《科学》(Science)杂志的年度重大科技突破。
1.2 Illumina测序仪
Illumina测序仪通常也被称作Solexa测序仪(Illumina测序仪的特点见表5)。它适用于采用各种方法制备的DNA文库,文库中DNA片段可以长达数百bp,并可通过桥式PCR来扩增模板片段(图5b)。在桥式PCR反应中,正向引物和反向引物都被通过一个柔性接头(flexible linker)固定在固相载体(solid substrate)上。经过PCR反应,所有的模板扩增产物就都被固定到了芯片上固定的位置。
值得注意的是,Illumina测序仪使用的桥式PCR与传统的桥式PCR有所不同,它会交替使用Bst聚合酶进行延伸反应以及使用甲酰胺(formamide)进行变性反应。这样,经过桥式PCR扩增之后,也会在固相载体上形成一个个的模板“克隆”。一块芯片的8条独立“泳道”上每一条泳道都可以容纳数百万的模板“克隆”,这样一次就可以同时对8个不同的文库进行测序。
经过上述PCR扩增步骤之后,所有的模板都被线性化处理(linearization)而形成单链模板,接着与测序引物退火、杂交。随后使用修饰的DNA聚合酶和四种核苷酸混合试剂进行单碱基延伸测序反应(图6b)。这些核苷酸试剂都经过两种方式处理过,它们都是可逆的终止子(reversible terminator)。这些核苷酸的3’羟基端都有一个可被化学法切除的基团,这样每一次反应都只会掺入一个核苷酸,同时每种核苷酸都标记上了可被化学法切除的不同颜色的荧光基团,以标识每种碱基。经过一轮单碱基掺入反应采集到信号之后,就可以通过化学方法切除上述被掺入核苷酸上标记的两个基团,然后就能够继续掺入下一个核苷酸,重复测序反应了。这种测序方法对36bp长度的序列测序准确率是非常高的,不过如果处理更长的序列,准确率就会有所降低了。
1.3 AB SOLiD测序仪
AB SOLiD测序仪可以对由任何方法制成的DNA文库进行测序。AB SOLiD测序仪有一个极大的特点就是能够将富集模板片段的微珠在芯片上进行高度可控的任意排列。AB SOLiD测序仪也是使用如图5a中所示的微乳液PCR方法扩增模板片段的,不过,它这里使用的是直径只有1μm的小磁珠。PCR扩增反应结束之后,微乳液滴被打破,小磁珠被富集起来固定到固态平板上,制成高密度测序芯片。后面的合成测序法由DNA连接酶而非DNA聚合酶完成。
首先,通用引物与模板片段两端的接头序列互补结合,然后连接酶将一个被荧光标记的8bp长的核酸探针片段(fluorescently labeled octamers)连接到引物末端(图6c)。这段8bp长的核酸探针片段是经过设计的,比如其中第五位碱基上就标记了荧光。连接反应完成之后,就可以采集荧光图像,然后在第五位碱基和第六位碱基之间切断,去掉荧光标签。如此反复,就可以获得每间隔四个碱基的第五号碱基的确切信息,比如第5号碱基、第10号碱基、第15号碱基以及第20号碱基等等。经过几轮这样的循环之后,已经获得延伸的引物会变性脱落,再重新结合上新的引物从头开始新一轮测序,不过这一次可能获得的是第4号碱基、第9号碱基、第14号碱基以及第19号碱基的信息。我们可以使用不同长度的引物(+1或者-1)或者使用在不同位点(比如第2号碱基)标记荧光的8bp核酸探针片段达到这个目的。如此反复,最终就能获得整条模板片段的完整序列信息。
AB SOLiD测序仪还有一个特点就是使用了双碱基编码技术(two-base encoding),该技术具有误差校正功能,因为它是通过两个碱基来对应一个荧光信号而不是传统的一个碱基对应一个荧光信号,这样每一个位点都会被检测两次,因此出错率明显降低。
Polonator测序仪是一个和AB SOLiD测序仪比较相似的产品,因为它也运用了J.S等人和哈佛大学Church研究小组开发的部分系统。Polonator测序仪同样也使用微乳液PCR法扩增模板片段,使用连接酶法测序。不过,Polonator测序仪的价格要比其它第二代测序仪低得多。而且更重要的是,Polonator测序仪是一个开源的设备,用户可以通过自己编程“设计”出最适合自己的测序仪。不过,Polonator测序仪目前可测序的长度还非常有限。
值得注意的是,454测序仪、SOLiD测序仪以及Polonator测序仪还都存在一个共同的不足,那就是微乳液PCR技术实在是太过麻烦并且对实验操作的技术要求较高。不过从另一方面来说,使用仅仅只有1μm大小的磁珠构成的高密度测序芯片进行测序(不论是使用聚合酶法、连接酶法,还是其它的生化方法)是最有可能实现的高通量测序方法。因为1μm是衍射技术(diffraction)所能分辨的极限大小了。另一方面,最近报道的使用1μm磁珠进行高分辨率芯片点样技术的突破,使我们有望实现每个测序模板一个像素(one pixel per sequencing feature)的愿望。
1.4 HeliScope测序仪
HeliScope测序仪是由Quake团队设计开发的,它实际上也是一种循环芯片测序设备。不过,HeliScope测序仪最大的特点是无需对测序模板进行扩增,它使用了一种高灵敏度的荧光探测仪直接对单链DNA模板进行合成法测序。首先,将基因组DNA切割成随机的小片段DNA分子,并且在每个片段末端加上poly-A尾。然后通过poly-A尾和固定在芯片上的poly-T杂交,将待测模板固定到芯片上,制成测序芯片。最后借助聚合酶将荧光标记的单核苷酸掺入到引物上(图6d)。采集荧光信号,切除荧光标记基团,进行下一轮测序反应,如此反复,最终获得完整的序列信息。根据最近的报道,经过数百轮这种单碱基延伸可以获得25bp或更长的测序长度。HeliScope测序仪的其它特点见表6。


原文检索:Jonathan M Rothberg & John H Leamon. (2008) The development and impact of 454 sequencing. Nature Biotechnology, 26(10): 1117-1124.
筱玥/编译
2. 用于处理新一代测序技术数据的软件和标准
各种新一代测序仪的飞速发展面临着一个极其重要的问题,那就是生物信息学问题,这些问题包括序列质量评分(sequence quality scoring)问题、序列比对问题、序列组装问题、数据发布问题等。下面将逐个进行讨论。
2.1 序列质量问题
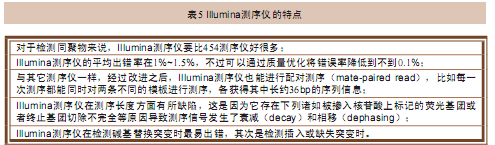
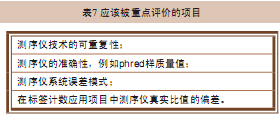
目前,序列质量评分问题是受到广泛关注的一个问题。造成这种现象的原因主要是因为所有新一代测序仪的测序质量都不高,而且不同的序列情况都有各自的误差率。随着新一代测序仪产品的不断成熟,在临床及科研工作中的应用范围越来越广,它们的测序质量也就变得重要起来,而且我们也需要对各个测序仪的测序质量有一个清晰的、可靠的评价标准。由于这个问题还只是刚刚出现,所以我们有机会设立一个全球统一的、标准化的评价体系对目前现有的以及将来即将出现的测序仪进行评价。我们希望避免再次发生类似过去几个芯片厂家之间进行数据比较的尴尬局面。对于测序仪的应用范围进行标准化的质量评价也是有好处的。比如评价从头测序的质量、评价测序结果与参考序列的相似度、评价测序仪发现突变以及多态性的能力以及对测序仪在进行大规模测序项目研究时的质量可靠性进行评价等。表7列出了几项应该被重点评价的项目。
这些质量数据都应该以一种简单、标准化的方式包含在测序结果中。现在所有的测序仪器生产商也都在他们的测序报告中加入了测序质量信息,消费者可以借此对数据进行交叉比较,甚至还有可能各取所长,将不同测序仪的测序结果整合起来,获得最佳的测序结果。目前,旨在从短片段测序结果中发现多态性以及突变位点的重测序项目经常会依靠“主要投票机制(majority voting scheme)”。该方法易于操作,但是容易出错,假阴性率较高。诸如Brockman小组和Quinlan小组开发的,更多更好的用于发现单核苷酸多态性的方法是将误差率与单个碱基信号联系起来,即误差率与测序质量和序列内容相关,这样就能获得更准确的结果。我们估计,像phred样质量值之类的评价体系以及“第三方”算法的不断发展,最终一定会诞生一个很好的发现多态性的工具。
2.2 用于分析数据的软件以及生物信息学工具
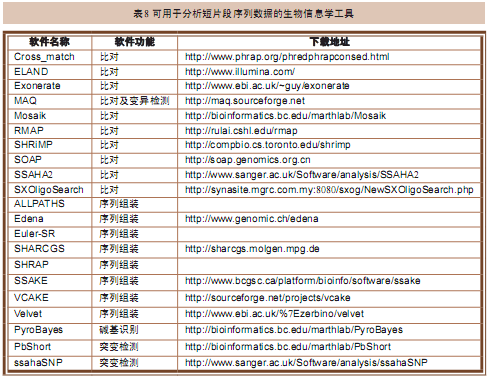
虽然这些软件和工具都还没有完全商业化,但我们还是能获得一些软件用来分析新一代测序仪的测序结果(表8)。这些软件和工具的功能主要集中在4个方面(表9)。
序列比对和组装是一个颇受关注的问题。虽然BLAST和BLAT等序列比对工具对于长片段的序列比对来说非常有用,但是它们不适合新一代测序仪获得的短片段序列。因此,有大量的针对短测序片段的比对工具出现,这些工具中允许错配或者空隙出现。这些工具中有些借鉴了非常成熟的序列比对算法,例如Smith-Waterman算法等,不过还是有很多新的专门针对短序列片段的算法出现。比如SOAP就是这样一种新型算法,这个软件包能非常有效地进行有空隙或者无空隙的序列比对。该软件包使用了一种内存密集种子(memory-intensive seed)算法和查表格(look-up table)算法来加快比对速度,同时能够对最容易出错的3’端序列进行反复调整。
其它用来加速比对过程的方法还包括“字节编码(bit encoding)”技术。该技术能将序列信息压缩成计算机信息,这样更利于计算机管理和分析。越来越多的比对软件在进行比对时开始考虑数据质量,比如能处理Solexa测序仪测序结果和SOLiD测序仪测序结果的MAQ软件,以及能用最新的“字符空隙转化成彩色空隙(color-space to letter-space)”的Smith-Waterman算法对SOLiD测序仪的双碱基编码测序结果进行分析的SHRiMP 工具(http://compbio.cs.toronto.edu/shrimp/)。

和序列比对算法的进展情况不同,如何将相对低质量的短片段序列从头组装起来还是一大难题。不过,已经有好几种组装工具开始投入使用了。对于这种短片段序列的从头组装工作来说,配对序列对于整个工作的成功与否影响最大,因此已经有人开始利用配对序列设计新的组装工具了。
2.3序列数据出版、发行、组织存档原则
目前对于如何组织、存档以及发布这些新一代测序仪产生的短片段序列结果正处于热烈的讨论之中,人们希望制定一个类似芯片试验(microarray experiments,http://uhts.lbl.gov/)时制定的MIAME(Minimum Information About a Microarray Experiment)规则。这些早期的工作经验在如何处理包括生物学注释信息、临床原始数据、关键试验细节(比如样品特征、样品处理方法)在内的元数据,以及如何处理、出版发行这些数据等方面给了我们良好的建议。如何对这些新一代测序仪的测序结果数据进行公共管理也是一个需要探讨的问题。NCBI最近专门为短片段序列建立了数据库Short Read Archive(SRA),并同步制定数据提交格式。SRA数据库不仅会收集包括实验注释信息、试验参数等信息的数据,而且还会被整合到Entrez查询系统当中。目前的工作主要包括开发线上搜索工具、数据图形化工具,这些工作有望在近期完成。
3. 新一代测序技术的前景
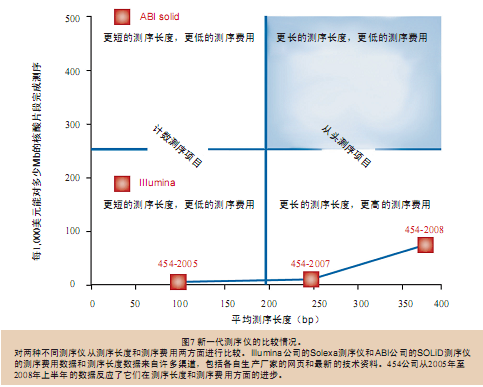
在2007年6月,James Watson的基因组序列登录到了GenBank数据库当中,这是第一次使用非Sanger测序法获得了人类个体基因组序列,并且第一次将个人基因组序列公之于众。整个测序过程在两个月之内就完成了,花费不到100万美元,这只占耗时10年之久的人类基因组计划使用经费的千分之一,同时还是2007年5月在网上公布结果的Venter基因组计划费用的百分之一。我们比较了454测序仪最初的技术参数(每次可以获得两千万碱基序列,测序长度100bp,准确率96%)和用于对James Watson进行测序时的技术参数(每次可以获得一亿碱基序列,测序长度250bp,准确率超过99%),结果发现摩尔定律真的适用于基因组测序领域。
454测序仪和其它的新一代测序仪(图7)一起,展示出了小型化技术和并行处理技术的威力,它们提高了处理通量,降低了测序费用。除了引领新一代测序技术的发展之外,454公司的研发团队还开发了体外DNA文库构建、模板扩增等技术,而且这些技术现在都已经被市场上其它新一代测序仪所广泛使用。很快,随着计算机技术的飞速发展,个体基因组测序的费用将会由100,000美元降低到10,000美元,继而降低到1,000美元甚至更低。
个人基因组时代马上就要到来了!
4. 新一代测序技术与传统测序技术的优缺点分析
从费用角度、适用范围和限制性来说,传统测序仪和新一代测序仪之间具有明显的差距。因此,对于每一个具体的项目来说,都需要仔细考虑,选择出最合适的测序仪。传统的Sanger测序法适用于对kb~mb长度的DNA片段进行的小规模的测序项目。Sanger测序法相比新一代测序法而言具有极大的“间隔尺寸(granularity)”,既能用于大型项目也能用于小型项目。虽然与传统测序仪相比,新一代测序仪在某些方面很明显地处于劣势,比如在测序长度和准确率方面,但即便如此,在处理大规模的测序项目时大家还是倾向于选择新一代测序仪。
看看新一代测序仪对以往使用传统测序仪进行的生殖细胞突变和体细胞突变研究的帮助就可以认识到它们的作用有多么强大。在这项研究里,使用Sanger测序法除了试剂这一项费用之外,其它的费用也远远高过了使用其它新一代测序仪。这些其它费用包括在96孔板或384孔板中处理样品的费用、电泳费用、大量的生物信息学处理费用以及设备维护人工费用等。研究人员最近对100份样品中的100个基因使用传统测序方法究竟需要花费多少费用进行了一次非正式的调查,假设每个基因平均由10个外显子组成,结果发现整体费用在30万美元至100万美元不等,价格依据测序单位是非盈利的基因组测序中心还是商业化的测序服务机构而不同。很显然,这么高昂的费用对于任何一个实验室来说都是难以承受的。新一代测序仪除了能将测序费用降低好几个数量级之外,它们还具有所需仪器设备少的优点,不过新一代测序仪在后续数据处理方面会碰到问题。
各款新一代测序仪之间也有非常明显的差异(表10),它们都有各自“拿手”的绝活(表11)。有一些测序项目,比如重测序(resequencing)对于测序仪的测序长度要求就没有从头测序的要求高。对于需要依靠标签计数(tag counting)的测序项目,例如在定量分析蛋白质与DNA之间的相互作用时,我们就会更加需要能将待测片段分割成尽量多、尽量小片段的测序方法。测序的准确度和各自相对拿手的项目,比如是善于发现插入、缺失突变还是善于发现碱基替换突变也是需要着重考虑的问题。另外,在进行从头测序或发现结构性变异的研究时使用的配对测序法已经广泛应用于各种新一代测序仪当中。这时,这些配对的模板片段在芯片上的分布情况,比如相互之间的距离远近等就是需要重点考虑的问题了。
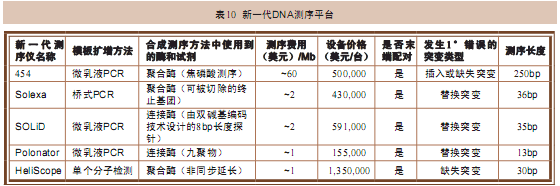
注:DNA测序领域的快速发展使得对各类测序方法的价格及读长的评估在很短时间内便失去意义。Roche Applied Science、Illumina及Applied Biosystems公司目前都在不断推出新的产品。表中列出的测序费用只是对使用的反应试剂费用的一个估算。测序长度指的是单链长度。
最后,需要考虑的当然是价格因素,各个新一代测序仪的费用都不相同,作为消费者,当然希望各个测序仪生产厂家之间的竞争更加激烈一点。单纯比较每个碱基的测序费用是一个不错的选择方法,不过有时这也会误导我们,比如准确率更高的方法当然费用会高一些。

5. 总结
过去几年间,新一代测序技术获得了突飞猛进的进展,同时有好几款使用大规模平行循环芯片测序技术的测序仪得到了广泛的应用。这几款测序仪虽然使用的技术有所差异,但是在测序数据的质量和数量方面都有着同样的特征,因此也都面临着同样的试验设计、数据分析和注释的问题。不过,这些新一代测序仪将以往的测序费用降低了好几个数量级。鉴于此,以前只有大型测序中心才能够开展的项目,现在在小型实验室里也能顺利进行了。由于新一代测序仪的出现,测序研究领域也开始升温,有些研究团队正在努力开发新的测序技术希望能够取代现有的新一代测序仪。按照目前的发展速度,我们很难估计几年之后的情况。不过,能够预计的是,下、下一代或者说是第三代测序仪一定会像十年前的芯片技术一样,迅速地普及开来,从而成为常规的技术。希望人们不仅关注测序技术本身的发展,更加关注如何利用测序技术来揭开生物学和医学上的众多谜团。
原文检索:
Jay Shendure & Hanlee Ji. (2008) Next-generation DNA sequencing. Nature Biotechnology, 26(10):1135-1145.
Jonathan M Rothberg & John H Leamon. (2008) The development and impact of 454 sequencing. Nature Biotechnology, 26(10): 1117-1124.
YORK&筱玥/编译
小词典
1. Polony芯片
该方法是由瑞士日内瓦雪兰诺制药研究中心的Eric Kawashima、俄罗斯科学院的Alexander Chetverin和当时在美国哈佛大学的密特拉共同开发的。他们制作出了可排列在载玻片或凝胶分子层上的一个个独立的聚合酶群落,成为polony,每一个聚合酶群落里都含有一个DNA模板分子,通过PCR反应就可以获得大量的模板。这些扩增后的聚合酶群落就像菌落一样一个个散布在载玻片或凝胶分子层上,成为polony芯片。每一个polony直径约为1μm,一个芯片上可以承载数十亿个聚合酶群落。
2. 元基因组(metagenomics)
元基因组又称环境基因组学,是指同时被研究的整个微生物群落的DNA。
3. Phred样质量值(Phred-like quality scores)
Phred样质量值最初是被定义用来衡量自动DNA测序质量的。Phred样质量值被广泛用来衡量DNA测序仪的测序质量好坏,并且被用来比较不同测序方法之间的效率高低。
